
(原文地址:The Nobel Prize in Physiology or Medicine 2016)
科学背景:自噬机制的发现
2016年诺贝尔生理学或医学奖授予大隅良典(Yoshinori Ohsumi),以表彰他在自噬机制方面所做的发现(for his discoveries of mechanisms for autophagy)。巨型自噬(“自我吞噬”,下文简称为自噬)是一种进化保守机制,通过这个过程,真核细胞可以通过将细胞质的一部分包裹在双层膜囊泡中并送往溶酶体进行消化,从而回收其自身的一部分内容。与其他细胞降解机制不同,自噬能够去除寿命较长的蛋白质、大型分子复合体和已经过时或损坏的细胞器。自噬在饥饿状态下介导细胞非必需部分的消化和回收,并参与多种生理过程,在这些过程中,细胞成分必须被移除,为新的成分腾出空间。此外,自噬是一种关键的细胞过程,能够清除入侵的微生物和有毒的蛋白质聚集体,因此在感染、衰老以及许多人类疾病的发病机制中发挥重要作用。尽管自噬在1960年代就已被发现,但其机制和生理学意义几十年来仍然了解甚少。大隅良典的工作极大地改变了我们对这一重要细胞过程的理解。1993年,大隅发布了他的开创性发现,确定了酿酒酵母中对自噬至关重要的15个基因。在一系列经典的后续研究中,他克隆了酵母和哺乳动物细胞中的这些基因中的几个,并阐明了它们编码蛋白质的功能。基于大隅良典的开创性发现,人们现在已经认识到自噬在人体生理学和疾病中的重要性。
神秘的自噬
20世纪50年代初,克里斯蒂安·德·迪夫(Christian de Duve)对胰岛素(insulin)的作用产生了兴趣,他使用阿尔伯特·克劳德(Albert Claude)开发的细胞分级(cell fractionation)方法研究了葡萄糖-6-磷酸酶(glucose-6-phosphatase)在细胞内的定位。在对照实验中,他还跟踪了酸性磷酸酶(acid phosphatase)的分布,但在新鲜分离的肝脏细胞分级样品中未能检测到任何酶活性。值得注意的是,如果将这些分级样品存放在冰箱中五天,酶活性就会重新出现 。事情很快就清楚了,蛋白水解酶被隔离在一种以前未知的膜结构内,德·迪夫将其命名为溶酶体(lysosome)
。通过将纯化的富含溶酶体的肝脏细胞样品与肝脏切片进行比较电子显微镜观察,确认溶酶体是一种独特的细胞器
。克里斯蒂安·德·迪夫、阿尔伯特·克劳德和乔治·帕拉德(George Palade)一起,因其关于细胞结构和功能组织的发现而荣获1974年诺贝尔生理学或医学奖。
溶酶体被发现不久后,研究人员发现,在小鼠正常肾脏发育过程中,细胞质的一部分会被隔离到膜结构内 。当发生水肿病时,大鼠肾小管近曲小管细胞中也会观察到类似的结构,其中包含少量细胞质和线粒体
。发现这些小泡在退化初期阶段与含酸性磷酸酶的颗粒共定位,并且随着退化的进展,这些结构的数量增加
。含有退化细胞质的膜结构也存在于正常大鼠肝细胞中,其数量在糖皮质激素灌流(glucagon perfusion)
或暴露于有毒试剂后
显著增加。
克里斯蒂安·德·迪夫认识到这些结构有能力消化细胞内部分内容物,于1963年首次提出“自噬”(autophagy)这个术语,并在几年后发表的一篇综述文章中广泛讨论了这一概念 。当时,基于电子显微镜研究的结果,已经有充分的理由证明哺乳动物细胞中存在自噬8。已知自噬在基础水平上低速进行,并在多种组织的分化和重塑过程中增加,包括大脑、肠道、肾脏、肺、肝脏、前列腺、皮肤和甲状腺
。人们推测,自噬可能是一种响应饥饿以应对代谢压力的机制
,并且可能在疾病发病机制中发挥作用
。此外,已经证实自噬发生在各种单细胞真核生物和多细胞生物中,例如变形虫、纤细裸藻(Euglena gracilis)、四膜虫(Tetrahymena)、昆虫和蛙类
,这指向了其在进化过程中的保守功能。
在接下来的几十年里,这个领域的进展有限。有报道称营养物质和激素能够影响自噬;氨基酸缺乏可诱导 ,而胰岛素刺激可抑制
哺乳动物组织中的自噬。一种小分子,3-甲基腺嘌呤(3-methyladenine),被证实可以抑制自噬
。有研究结合了细胞分级、放射自显影和电子显微镜等技术,提供了自噬早期阶段包括双膜结构——吞噬泡(phagophore)形成的证据,该结构扩展包围了一部分细胞质并闭合成一个不含水解酶的囊泡——自噬体(autophagosome)
(图1)。
尽管有许多迹象表明自噬可能是一个重要的细胞过程,但其机制和调控仍然不清楚。只有少数几个实验室在研究这个问题,主要采用相关性或描述性的方法,关注自噬的后期阶段,即与溶酶体融合前后的过程。我们现在知道,自噬体是短暂存在的,其存在时间仅大约10-20分钟,然后就与溶酶体融合,这使得其形态学和生化研究非常困难。
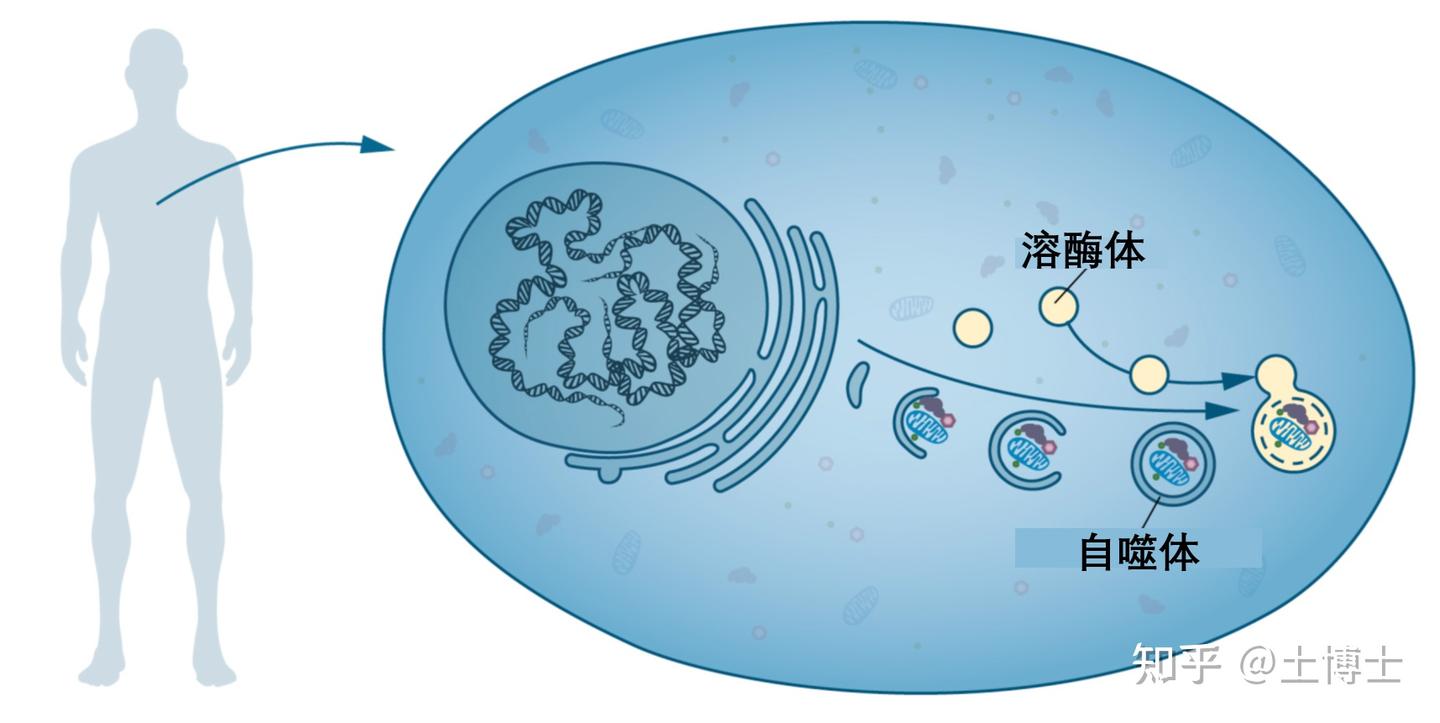
到了20世纪90年代初,距离德·迪夫首次提出自噬这一术语近30年后,这一过程仍然是一个生物学谜团。由于没有可用的分子标记,自噬机制的组分仍然难以捉摸。许多基本的问题仍然没有答案:自噬过程是如何启动的?自噬体是如何形成的?自噬对于细胞和机体的生存来说有多重要?自噬在人类疾病中有何作用?
自噬机制的发现
20世纪90年代初,大隅良典(Yoshinori Ohsumi)当时是东京大学的助理教授,决定使用出芽酵母Saccharomyces cerevisae作为模型来研究自噬。他首先要解决的问题是这种单细胞生物体内是否存在自噬。酵母的液泡(vacuole)是哺乳动物溶酶体的功能等同物。大隅推理说,如果酵母中存在自噬,抑制液泡酶类将导致被吞噬的细胞质成分在液泡中积累。为了验证这一假设,他建立了缺失液泡蛋白酶A(proteinase A)、蛋白酶B(proteinase B)和羧肽酶(carboxy-peptidase)的酵母株 。他发现,当改良后的酵母在缺乏营养的培养基中生长时,自噬体在液泡中积累,产生了在光学显微镜下可见的异常液泡。他找到了一种独特的表型,可以用来发现控制自噬启动的基因。通过在缺乏液泡蛋白酶的酵母细胞中诱发随机突变,大隅发现了第一个无法在液泡中积累自噬体的突变体
;他将这个基因命名为autophagy 1(APG1)。随后,他发现APG1突变体在缺少氮源的培养基中存活能力相比野生型酵母快速下降。作为第二种筛选方法,他利用这一更便捷的表型和更多的表征方法,识别出了75个可被划分为不同互补组的隐性突变体。在1993年发表在FEBS Letters杂志上的一篇文章中,大隅报告了他对真核细胞中激活自噬所必需的多达15个基因的发现
。他将这些基因命名为APG1-15。随着酵母和其他物种中新的自噬基因被识别出来,普遍采纳了使用ATG缩写的统一基因命名系统
,此后本文将使用这一命名。
在随后的几年中,大隅克隆出了几个ATG基因 ,并描述了其蛋白质产物的功能。ATG1基因的克隆揭示了它编码一个丝氨酸/苏氨酸激酶(serine/threonine kinase),证明了蛋白质磷酸化在自噬中的作用
。其他的研究表明,Atg1与ATG13基因的产物形成复合体,且这种相互作用受到雷帕霉素靶蛋白(target of rapamycin,TOR)激酶的调控
。在营养丰富条件下生长的细胞中,TOR处于活跃状态,对Atg13进行过度磷酸化,从而阻止了Atg13:Atg1复合体的形成。相反,在饥饿条件下TOR被失活,去磷酸化的Atg13与Atg1结合,激活自噬
。随后研究表明,活性激酶实际上是一个五聚复合体(pentameric complex)
,除了Atg1和Atg13外,还包括Atg17、Atg29和Atg31。这个复合体的组装是形成自噬体所需的一系列事件中的第一步。
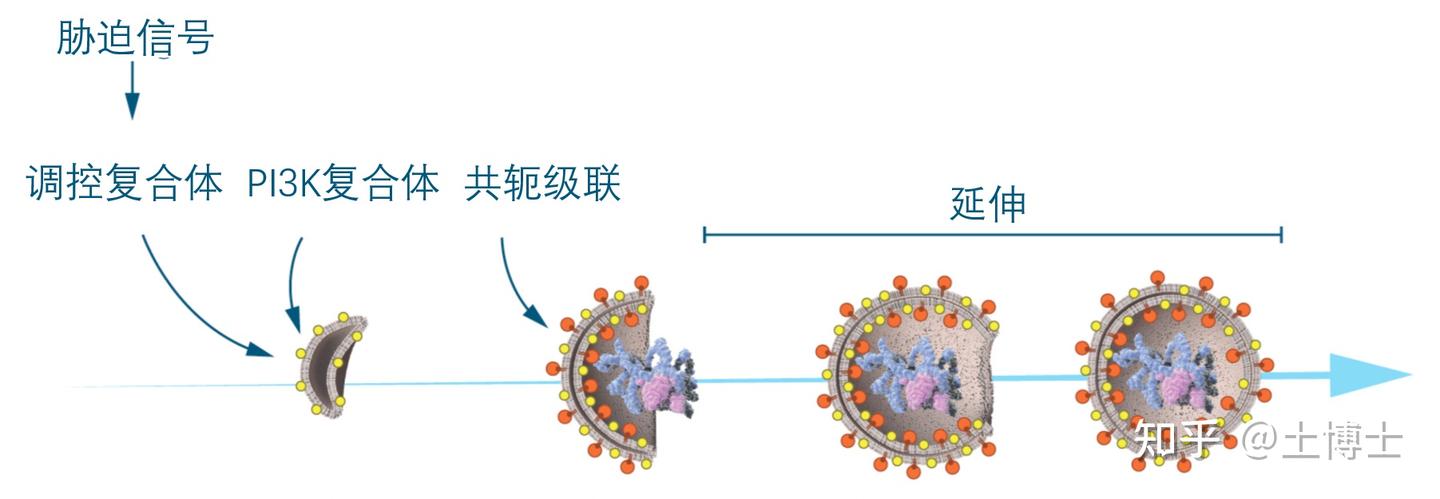
自噬体的形成涉及到一种内在膜蛋白Atg9,以及一个磷酸肌醇-3-激酶(phosphatidylinositol-3 kinase,PI3K)复合体 ,该复合体由液泡蛋白分选相关蛋白34 (vacuolar protein sorting-associated protein 34,Vps34)、Vps15、Atg6和Atg14组成。这个复合体生成磷酸肌醇-3-磷酸(phosphatidylinositol-3 phosphate),随后其他的Atg蛋白质被招募到膜结构上。膜扩展以形成成熟的自噬体涉及两个泛素样蛋白(ubiquitin-like protein)连接的级联反应(图2)。
关于Atg8定位的研究显示,在生长中的酵母细胞的细胞质中,该蛋白质均匀分布,而在饥饿的细胞中,Atg8形成大的聚集体,与自噬体和自噬体内部的物质共定位 。大隅良典意外地发现,Atg8的膜定位依赖于两个泛素样连接系统(ubiquitin-like conjugation system),这两个系统依次作用,促进Atg8与膜脂磷脂酰乙醇胺(phosphatidylethanolamine)的共价结合。这两个系统共享相同的激活酶Atg7。在第一个连接事件中,Atg12通过与Atg7的半胱氨酸残基形成硫酯键而被激活,然后转移给连接酶Atg10,该酶催化它与Atg5蛋白质的共价结合
。进一步的研究表明,Atg12:Atg5共价体招募了Atg16,形成一个三分子复合体,在自噬过程中发挥着关键作用,作为第二个泛素样连接系统的连接酶
。在这个独特的第二个反应中,Atg8的C末端精氨酸被Atg4去除,随后成熟的Atg8由Atg7激活,并转移给Atg3连接酶
。最终,因为Atg12:Atg5:Atg16连接酶促进Atg8与磷脂酰乙醇胺的连接,这两个连接系统汇聚
。脂化的Atg8是自噬体延伸和融合的关键驱动力
。这两个连接系统在酵母和哺乳动物之间高度保守。在哺乳动物系统中,广泛使用与酵母Atg8同源的哺乳动物Atg8(即轻链3,light chain 3,LC3)的荧光标记,作为自噬体形成的标志物
。
大隅良典及其同事是第一批鉴定酵母ATG基因的哺乳动物同源基因的科学家,这使得人们能够研究自噬在更高级真核生物中的功能。不久后,遗传学研究显示,缺乏ATG5基因的小鼠在出生时看起来是正常的,但由于无法应对出生前的饥饿状态,它们在出生后的第一天就会死亡 。通过对缺失自噬机制不同组件敲除小鼠模型的研究,证实了这一过程在多种哺乳动物组织中的重要性
。
大隅良典的开创性研究引发了对自噬的巨大兴趣。自2000年代初以来,这一领域已经成为生物医学研究中最为密集研究的领域之一,相关的出版物数量显著增加。
自噬的不同类型
继大隅良典的开创性发现之后,现在可以根据被降解的内容物来区分不同类型的自噬。最为广泛研究的自噬形式——宏自噬(macroautophagy),能够降解大部分的细胞质和细胞器。非选择性自噬(non-selective autophagy)持续进行,并能在响应压力(例如饥饿)时得到有效诱导。此外,特定类别底物(蛋白聚集体、胞质细胞器或入侵的病毒和细菌)的选择性自噬(selective autophagy)涉及到特定的衔接蛋白(adaptor),这些衔接蛋白能识别内容物并将其定位到自噬体膜上的Atg8/LC3处 。自噬的其他形式包括微自噬(microautophagy)
,其涉及到通过溶酶体膜的内折叠直接吞噬细胞质组分,以及伴侣介导的自噬(chaperone-mediated autophagy,CMA)。在CMA中,带有特定识别信号的蛋白质通过与伴侣复合体(chaperone complex)的结合直接转运进入溶酶体
。
自噬在健康和疾病中的作用
自噬的分子特征研究提供的基础数据在推进对这一过程及其在细胞生理学和多种病理状态中的作用的理解方面发挥了重要作用(见图3)。自噬最初被认识为对胁迫的细胞响应,但现在我们知道,该系统在基础水平上持续运作。与优先降解短半衰期蛋白质的泛素-蛋白酶体系统(ubiquitin-proteasome system)不同,自噬能够移除长半衰期蛋白质,并且是唯一能够处理整个细胞器的过程,如线粒体、过氧化物酶体和内质网。因此,自噬在维持细胞稳态中发挥着重要作用。此外,自噬参与了多种生理过程,如需要清除大量胞质的细胞分化和胚胎发生。自噬在响应不同类型胁迫时的快速诱导,体现了其在对抗细胞损伤和许多与衰老相关疾病中的细胞保护功能和能力。
由于自噬流(autophagic flux)的失调直接或间接地涉及到广泛的人类疾病,自噬成为治疗干预的一个特别有希望的目标。关于自噬在疾病中作用的重要作用体现之一,是观察到Beclin-1(BECN1基因的产物)在大量人类乳腺癌和卵巢癌中发生突变。BECN1是酵母ATG6的同源物,它调节自噬过程的启动步骤 。这一发现为科学家们研究自噬在癌症中的作用上产生了极大的兴趣
。
错误折叠的蛋白质倾向于形成对细胞有毒的不溶性聚集体(aggregate)。为了应对这一问题,细胞依赖于自噬 。在神经退行性疾病(neurodegenerative disease)的果蝇和小鼠模型中,通过抑制TOR激酶来激活自噬,从而减少蛋白质聚集体的毒性
。此外,在小鼠大脑中,通过组织特异性地破坏Atg5和Atg7来失活自噬,会导致神经退行性疾病
。几种常染色体隐性遗传人类疾病(autosomal recessive human disease)表现出自噬受损,其特征是脑部畸形、发育迟缓、智力障碍、癫痫、运动障碍和神经退行
。
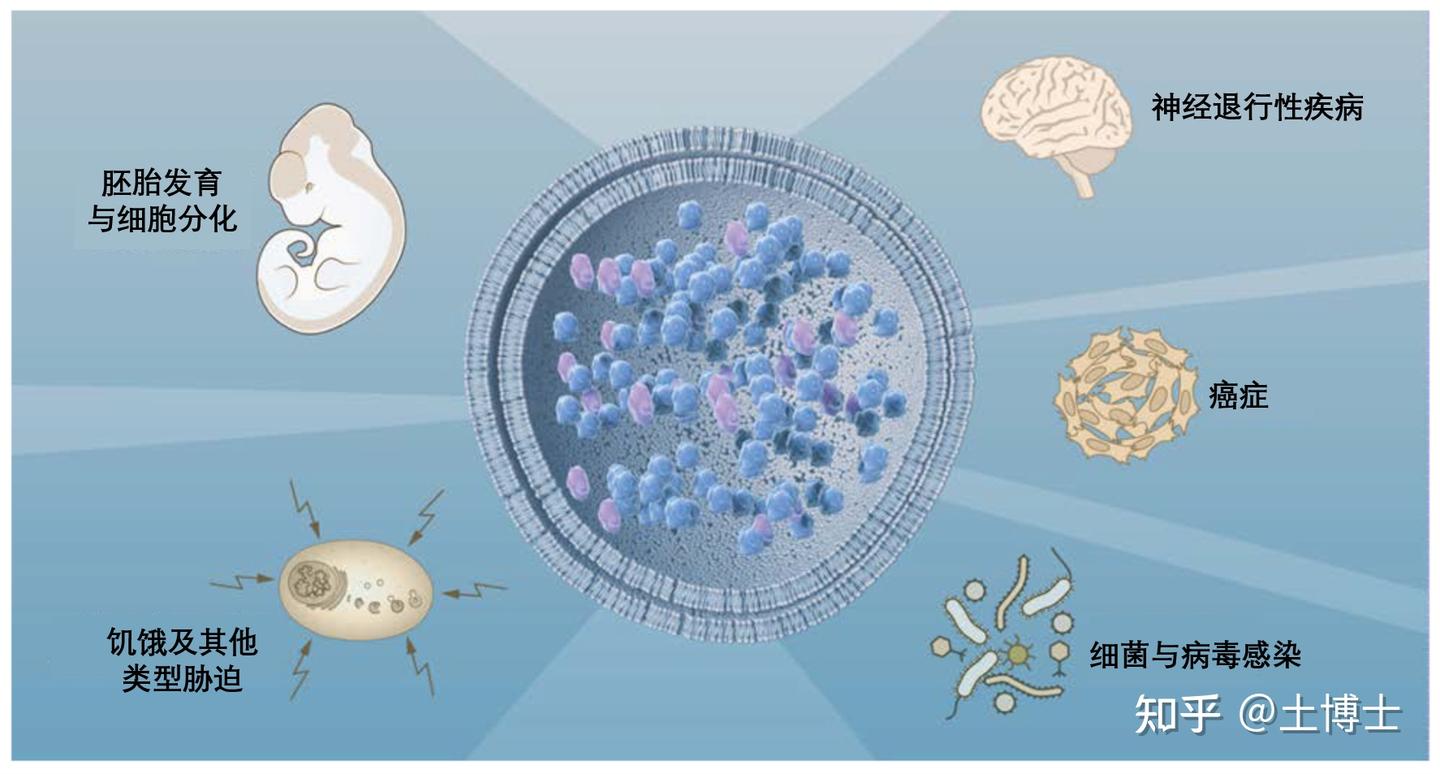
自噬消除侵入微生物的能力,这一现象被称为异噬(xenophagy),突显了其在激活免疫反应和控制传染病中的关键作用 。病毒和细胞内细菌已经发展出复杂的策略来规避这一细胞防御机制。此外,微生物还可以利用自噬来维持它们自身的生长。
结论
通过大隅良典对自噬基因的发现,以及对自噬分子机制的阐释,我们在理解细胞是如何回收其内容物方面迎来了一个新的范式。由于他的开创性工作,自噬被认识为细胞生理中的一个基本过程,对人类健康和疾病有着重大的影响。
尼尔斯-戈兰·拉尔松(Nils-Göran Larsson)医学博士哲学博士
线粒体遗传学教授,卡罗林斯卡学院
诺贝尔委员会兼职成员
诺贝尔大会成员
玛丽亚·G.马苏奇(Maria G. Masucci),医学博士哲学博士
病毒学教授,卡罗林斯卡学院
诺贝尔委员会兼职成员
诺贝尔大会成员
插图:马提亚斯·卡尔伦(Mattias Karlén)
参考文献
1. de DuveC. (2005). The lysosome turns fifty. Nat Cell Biol 7847–849.
2. de DuveC.PressmanB.C.GianettoR.WattiauxR.and AppelmansF. (1955) Tissue fractionation studies. 6. Intracellular distribution patterns of enzymes in rat-liver tissue. Biochem J 60604–617.
3. NovikoffA.BBeaufayH.and de DuveC. (1956) Electron microscopy of lysosome-rich fractions from rat liver. Journal Biophys Biochem Cytol. 2179–190.
4. ClarkS.L. (1957) Cellular differentiation in the kidneys of newborn mice studied with the electron microscope. J Biophys Biochem Cytol 3349–376.
5. NovikoffA.B. (1959) The proximal tubule cell in experimental hydronephrosis. J Biophys Biochem Cytol 6136–138.
6. AshfordT.P.and PorterK.R. (1962) Cytoplasmic components in hepatic cell lysosomes. J Cell Biol 12198–202.
7. NovikoffA.B.and EssnerE. (1962) Cytolysomes and mitochondrial degeneration. J Cell Biol 15140–146.
8. de DuveC.and WattiauxR. (1966) Functions of lysosomes. Annu Rev Physiol 28435–492.
9. BehnkeO. (1963) Demonstration of acid phosphatase-containing granules and cytoplasmic bodies in the epithelium of foetal rat duodenum during certain stages of differentiation. J Cell Biol18251–265.
10. BruniC.and PorterK.R. (1965) The fine structure of the parenchymal cell of the normal rat liver: I. General observations. Am J Pathol 46691–755.
11. HrubanZ.SpargoB.SwiftH.WisslerR.W.and KleinfeldR.G. (1963) Focal cytoplasmic degradation. Am J Pathol 42657–683.
12. MoeH.and BehnkeO. (1962) Cytoplasmic bodies containing mitochondriaribosomesand rough surfaced endoplasmic membranes in the epithelium of the small intestine of newborn rats. J Cell Biol 13168–171.
13. NapolitanoL. (1963) Cytolysomes in metabolically active cells. J Cell Biol 18478–481.
14. BonnevilleM.A. (1963) Fine structural changes in the intestinal epithelium of the bullfrog during metamorphosis. J Cell Biol 18579–597.
15. MortimoreG.E.and SchworerC.M. (1977) Induction of autophagy by amino-acid deprivation in perfused rat liver. Nature 270174–176.
16. PfeiferU.and Warmuth-MetzM. (1983) Inhibition by insulin of cellular autophagy in proximal tubular cells of rat kidney. Am J Physiol 244E109-114.
17. SeglenP.O.and GordonP.B. (1982) 3-Methyladenine: specific inhibitor of autophagic/lysosomal protein degradation in isolated rat hepatocytes. Proc Natl Acad Sci USA 791889–1892.
18. ArstilaA.U.and TrumpB.F. (1968) Studies on cellular autophagocytosis. The formation of autophagic vacuoles in the liver after glucagon administration. Am J Pathol 53687–733.
19. TakeshigeK.BabaM.TsuboiS.NodaT.and OhsumiY. (1992) Autophagy in yeast demonstrated with proteinase-deficient mutants and conditions for its induction. J Cell Biol 119301–311.
20. TsukadaM.and OhsumiY. (1993) Isolation and characterization of autophagy-defective mutants of Saccharomyces cerevisiae. FEBS Lett 333169–174.
21. KlionskyD.J.CreggJ.M. DunnW.A. Jr.EmrS.D.SakiaJ.SandovalI.V.SibirnyaY.A.SubramaniS.ThummM.VeenhuisM.and OhsumiY. (2003) A unified nomenclature for yeast autophagy-related genes. Dev Cell 5539-545.
22. KametakaS.MatsuuraA.Wada Y.and OhsumiY. (1996) Structural and functional analyses of APG5a gene involved in autophagy in yeast. Gene 178139-43.
23. FunakoshiT.MatsuuraA.NodaT.Ohsumi Y. (1997) Analyses of APG13 gene involved in autophagy in yeastSaccharomyces cerevisiae. Gene. 192207-213.
24. MatsuuraA.TsukadaM.WadaY.and OhsumiY. (1997) Apg1pa novel protein kinase required for the autophagic process in Saccharomyces cerevisiae. Gene 192245–250.
25. KamadaY.FunakoshiT.ShintaniT.NaganoK.OhsumiM.and OhsumiY. (2000) Tor-mediated induction of autophagy via an Apg1 protein kinase complex. J Cell Biol 1501507–1513.
26. OhsumiY. (2014) Historical landmarks of autophagy research. Cell Res 249–23.
27. KirisakoT.BabaM.IshiharaN.MiyazawaK.OhsumiM.YoshimoriT.NodaT.and OhsumiY. (1999) Formation process of autophagosome is traced with Apg8/Aut7p in yeast. J Cell Biol 147435–446.
28. MizushimaN.NodaT.YoshimoriT.TanakaY.IshiiT.GeorgeM.D.KlionskyD.J.OhsumiM.and OhsumiY. (1998) A protein conjugation system essential for autophagy. Nature 395395–398.
29. ShintaniT.MizushimaN.OgawaY.MatsuuraA.NodaT.and OhsumiY. (1999) Apg10pa novel protein-conjugating enzyme essential for autophagy in yeast. EMBO J 185234–5241.
30. MizushimaN.NodaT.and OhsumiY. (1999) Apg16p is required for the function of the Apg12p-Apg5p conjugate in the yeast autophagy pathway. EMBO J 183888–3896.
31. IchimuraY.KirisakoT.TakaoT.SatomiY.ShimonishiY.IshiharaN.MizushimaN.TanidaI.KominamiE.OhsumiM.et al. (2000) A ubiquitin-like system mediates protein lipidation. Nature 408488–492.
32. HanadaT.NodaN.N.SatomiY.IchimuraY.FujiokaY.TakaoT.InagakiF.and OhsumiY. (2007) The Atg12-Atg5 conjugate has a novel E3-like activity for protein lipidation in autophagy. J Biol Chem 28237298–37302.
33. NakatogawaH.IchimuraY.and OhsumiY. (2007) Atg8a ubiquitin-like protein required for autophagosome formationmediates membrane tethering and hemifusion. Cell 130165–178.
34. Xie Z.Nair U.Klionsky D.J. (2008) ATG8 controls phagophore expansion during autophagosome formation. Mol Cell Biol 193290-3298.
35. KabeyaY.MizushimaN.UenoT.YamamotoA.KirisakoT.NodaT.KominamiE.OhsumiY.and YoshimoriT. (2000) LC3a mammalian homologue of yeast Apg8pis localized in autophagosome membranes after processing. EMBO J 195720–5728.
36. MizushimaN.YamamotoA.MatsuiM.YoshimoriT.and OhsumiY. (2004) In vivo analysis of autophagy in response to nutrient starvation using transgenic mice expressing a fluorescent autophagosome marker. Mol Biol Cell 151101–1111.
37. KumaA.HatanoM.MatsuiM.YamamotoA.NakayaH.YoshimoriT.OhsumiY.TokuhisaT.and MizushimaN. (2004) The role of autophagy during the early neonatal starvation period. Nature 4321032–1036.
38. MizushimaN.and KomatsuM. (2011) Autophagy: Renovation of cells and tissues. Cell 147728-741.
39. LiuL.SakakibaraK.ChenQ.OkamotoK. (2014) Receptor-mediated mitophagy in yeast and mammalian systems. Cell Res 24787-795.
40. LiW.W.LiJ.BaoJ.K. (2012) Microautophagy: lesser-known self-eating. Cell Mol Life Sci 691125-1136.
41. CuervoA.M.and WongE. (2014) Chaperone-mediated autophagy: roles in disease and aging. Cell Res 2492–104.
42. LiangX.H.JacksonS.SeamanM.BrownK.KempkesB.HibshooshH.and LevineB. (1999) Induction of autophagy and inhibition of tumorigenesis by beclin 1. Nature 402672–676.
43. ChoiA.M.K.RyterS.W.and LevineB. (2013) Autophagy in human health and disease. N Engl J Med 368651–662.
44. RavikumarB.VacherC.BergerZ.DaviesJ.E.LuoS.OrozL.G.ScaravilliF.EastonD.F.DudenR.O'KaneC.J.et al. (2004) Inhibition of mTOR induces autophagy and reduces toxicity of polyglutamine expansions in fly and mouse models of Huntington disease. Nat Genet 36585–595.
45. RavikumarB.DudenR.and RubinszteinD.C. (2002) Aggregate-prone proteins with polyglutamine and polyalanine expansions are degraded by autophagy. Hum Mol Genet 111107–1117.
46. KomatsuM.WaguriS.ChibaT.MurataS.IwataJ.-I.TanidaI.UenoT.KoikeM.UchiyamaY.KominamiE.et al. (2006) Loss of autophagy in the central nervous system causes neurodegeneration in mice. Nature 441880–884.
47. HaraT.NakamuraK.MatsuiM.YamamotoA.NakaharaY.Suzuki-MigishimaR.YokoyamaM.MishimaK.SaitoI.OkanoH.et al. (2006) Suppression of basal autophagy in neural cells causes neurodegenerative disease in mice. Nature 441885–889.
48. Ebrahimi-FakhariD.SaffariA.WahlsterL.LuJ.ByrneS.HoffmannG.F.JungbluthH.and SahinM. (2016) Congenital disorders of autophagy: an emerging novel class of inborn errors of neuro-metabolism. Brain 139317–337.
49. NakagawaI.AmanoA.MizushimaN.YamamotoA.YamaguchiH.KamimotoT.NaraA.FunaoJ.NakataM.TsudaK.et al. (2004) Autophagy defends cells against invading group A Streptococcus. Science 3061037–1040.
50. GutierrezM.G.MasterS.S.SinghS.B.TaylorG.A.ColomboM.I.and DereticV. (2004) Autophagy is a defense mechanism inhibiting BCG and Mycobacterium tuberculosis survival in infected macrophages. Cell 119753–766.
术语表
- 溶酶体(lysosome):真核细胞质中含有分解酶的细胞器,这些酶被膜结构包裹着。
- 吞噬泡(phagophore):在巨型自噬初始阶段形成的囊泡。通过自噬机制,吞噬泡被延展,以吞噬细胞质成分。
- 自噬体(autophagosome):一种将细胞质的一部分包裹在双层膜中并与溶酶体融合的细胞器,其中的内容物随后被分解。自噬体是巨型自噬中的关键结构。
- 选择性自噬(selective autophagy):一种介导特定细胞质成分降解的巨型自噬类型。不同形式的选择性自噬包括线粒体自噬(mitophagy,降解线粒体)、核糖体自噬(ribophagy,降解核糖体)、脂肪滴自噬(lipophagy,降解脂肪滴)和异噬(xenophagy,降解侵入的微生物)等。